Ciencia de redes: el campo oculto detrás del aprendizaje automático, la economía y la genética de la que nunca habrás oído hablar (probablemente): una entrevista con el Dr. Aaron Clauset [Parte 1]
Por Amy Hodler, Gerente del Programa de Analítica
Blog Neo4j
Recientemente tuve la oportunidad de combinar trabajo y placer y reunirme con el Dr.
Aaron Clauset, un experto en ciencia de redes, ciencia de datos y sistemas complejos. En 2016, Clauset ganó el Premio Erdos-Renyi en Network Science, pero es posible que esté más familiarizado con su investigación anterior sobre leyes eléctricas, predicción de enlaces y modularidad.
El Dr. Clauset dirige el grupo de investigación que desarrolló la referencia del
conjunto de datos ICON (si está buscando datos de red para evaluar, marque esto ahora) y ha publicado recientemente investigaciones que arrojan luz sobre posibles conceptos erróneos sobre estructuras de red. Cuando apareció un viaje de negocios de última hora a Denver, hice el viaje a Boulder, donde Clauset es profesor asistente de ciencias de la computación en la Universidad de Colorado en Boulder.

El Dr. Aaron Clauset es Profesor Asistente de Ciencias de la Computación en la Universidad de Colorado en Boulder y en el Instituto BioFrontiers. También es parte de la facultad externa en el Instituto Santa Fe (para estudios de complejidad).
Entre el almuerzo y la próxima clase de Clauset, hablamos sobre la investigación reciente de su grupo y la dirección general de la ciencia de la red, y me fui con una superposición de desilusión y entusiasmo. El Laboratorio Clauset ha estado trabajando para ampliar la diversidad y el rigor de estudiar sistemas complejos y, al hacerlo, pueden desmantelar algunas creencias que datan de los años 90. (Debería haber sabido que no sería simple, estamos hablando de sistemas complejos después de todo).
Esto le importa a la comunidad gráfica de
Neo4j porque cualquier persona que analice las redes, especialmente si buscan atributos globales, necesita comprender la estructura y la dinámica subyacente. A continuación se encuentra un resumen de nuestra discusión.
¿En qué tipo de trabajo se enfoca su equipo?
Clauset: Mi grupo de investigación en CU Boulder actualmente incluye cinco Ph.D. estudiantes, junto con algunos maestros y varios estudiantes universitarios. Nuestra investigación se centra tanto en el desarrollo de nuevos métodos computacionales para la comprensión de conjuntos de datos complicados y desordenados, como en la aplicación de estos métodos para resolver problemas científicos reales, principalmente en entornos biológicos y sociales.
En el grupo, todos están involucrados en la investigación de alguna manera. Por ejemplo, el sitio web ICON (índice de redes complejas) fue construido por un par de estudiantes de licenciatura para aprender conceptos de redes y explorar herramientas.
Las redes son una de nuestras áreas de trabajo clave. Las redes son en realidad solo una representación, una herramienta para comprender sistemas complejos. Representamos cómo funciona un sistema social al pensar en las interacciones entre pares de personas. Al analizar la estructura de esta representación, podemos responder preguntas sobre cómo funciona el sistema o cómo se comportan las personas dentro de él. En este sentido, la ciencia de la red es un conjunto de herramientas técnicas que se pueden aplicar a casi cualquier dominio.
Las redes también actúan como un puente para comprender cómo las interacciones y dinámicas microscópicas pueden conducir a regularidades globales o macroscópicas. Pueden hacer un puente entre lo micro y lo macro porque representan exactamente qué cosas interactúan entre sí. Solía ser común suponer que todo interactúa con todo, y sabemos que eso no es verdad; en genética, no todos los pares de personas y no todos los pares de genes interactúan entre sí.

Tomado de “
Hierarchical structure and the prediction of missing links in networks“
Un esfuerzo extremadamente importante en la ciencia de redes es descubrir cómo la estructura de una red da forma a la dinámica de todo el sistema. En los últimos 15 años hemos aprendido que para muchos sistemas complejos, la red es increíblemente importante para configurar lo que les sucede a los individuos dentro de la red y cómo evoluciona todo el sistema.
El trabajo de mi grupo se centra en caracterizar la estructura de estas redes para que podamos comprender mejor cómo la estructura finalmente da forma a la función.
¿Hay puntos en común entre los diferentes tipos de redes?
Clauset: A fines de la década de 1990 y principios de la década de 2000, mucha energía en la conducción de la ciencia de redes provino de los físicos, que aportaron nuevas herramientas matemáticas, modelos y muchos datos nuevos. Una idea que popularizaron fue la hipótesis de que los patrones "universales" ocurrieron en redes de todo tipo: redes sociales, biológicas, tecnológicas, de información e incluso económicas, y que fueron impulsadas por un pequeño número de procesos fundamentales.
Este tipo de idea era bastante normal en una parte de la física. Por ejemplo, hay un modelo matemático universal de cómo funciona un imán que hace predicciones notablemente precisas sobre imanes reales de todo tipo.
El sueño de las redes era mostrar que lo mismo se podía hacer por ellos: que todos los diferentes tipos de redes podían explicarse mediante un pequeño conjunto de principios o procesos matemáticos básicos, o que caían en un pequeño número de categorías estructurales generales. Es una idea bastante poderosa e inspiró tanto un trabajo multidisciplinario realmente bueno como varias afirmaciones altamente provocativas.

La validez de algunas de las afirmaciones más audaces ha sido difícil de evaluar empíricamente porque requirió el uso de un conjunto grande y diverso de redes del mundo real para probar la "universalidad" empírica del patrón. Reunir un conjunto de datos de este tipo es parte de lo que nos llevó a armar el
índice de redes complejas, lo que llamamos el índice ICON.
Aunque todavía estamos expandiéndolo, mi grupo ya ha comenzado a revisar muchas de las afirmaciones iniciales sobre patrones universales en redes, incluida la idea de que "todas las redes están libres de escalas", o que solo las redes sociales tienen una alta densidad triangular, o que las redes se agrupan en "superfamilias" basadas en el patrón de su estructura local. Sorprendentemente, muchas afirmaciones sobre la estructura de las redes se han repetido una y otra vez en la literatura, pero no han sido escrutadas cuidadosamente con datos empíricos.
Resulta que muchos de estos patrones universales se desmoronan cuando se puede mirar a través de una gran variedad de redes. El reciente artículo de Kansuke Ikehara [
Characterizing the structural diversity of complex networks across domains] plantea una pregunta simple: si etiqueto una gran cantidad de redes de dónde provienen (por ejemplo, una red de transporte / carretera, una red social / en línea o una red metabólica / biológica) ¿puede utilizar el aprendizaje automático para descubrir qué características distinguen a estas clases de redes?
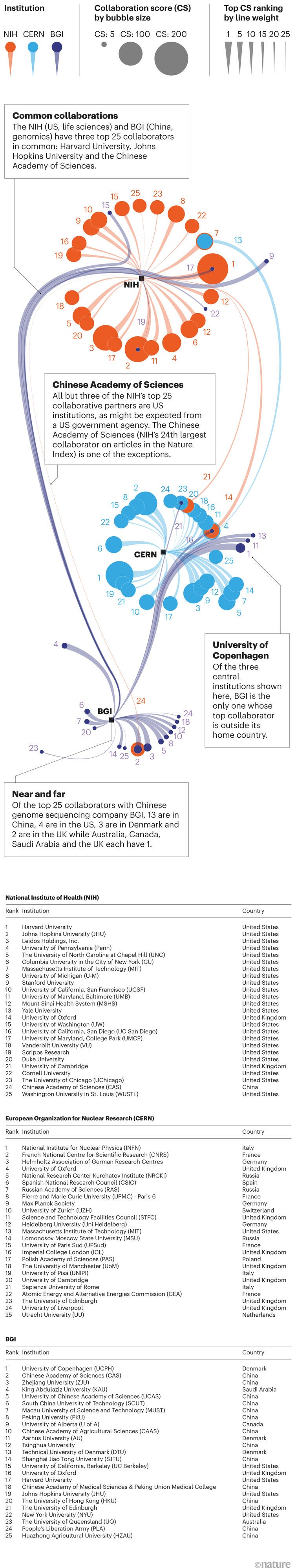
La diversidad estructural de las redes complejas. Si hay algunas "familias" de estructuras de red, entonces ningún algoritmo debería ser capaz de aprender a distinguir las diferentes redes dentro de una familia. En cambio, lo que encontramos fue que prácticamente todas las clases de redes se distinguían fácilmente de las demás clases.

Las redes sociales se agrupan en una parte del espacio de características, las redes biológicas generalmente están bien separadas de aquellas, etc., y esto es cierto para cada clase de red que examinamos. El claro mensaje para llevar a casa es que hay mucha más diversidad en las estructuras de red de lo que pensamos hace 20 años, y por lo tanto, mucho más trabajo por hacer para comprender de dónde viene esta diversidad.
La investigación de Ikehara reveló la diversidad estructural oculta de las redes y sugiere que puede haber menos patrones universales de lo que alguna vez se pensó. Al mismo tiempo, algunos grupos de redes están más cerca unos de otros en términos de su estructura.
Por ejemplo, encontramos que las redes de distribución de agua exhiben firmas estructurales similares a las redes miceliales fúngicas, lo que sugiere que pueden estar formadas por procesos subyacentes similares o problemas de optimización. De esta forma, el aprendizaje automático puede ayudarnos a identificar semejanzas estructurales y, por lo tanto, a ayudarnos a descubrir, de una manera basada en datos, dónde es más probable que encontremos una explicación mecánica común.
¿Cómo está evolucionando la ciencia de la red?
Clauset: En muchos sentidos, la ciencia de redes hoy se está diversificando y expandiendo. Esta expansión permite una gran especialización, pero hay una compensación. Ahora las personas pueden tomar métodos de red y aplicarlos en preguntas realmente específicas sobre sistemas realmente específicos.
Esto es enormemente productivo y un logro emocionante para la ciencia de redes. Pero, el crecimiento del trabajo disciplinario alrededor de las redes también significa que hay relativamente menos trabajo que cruza los límites disciplinarios. Sin espacios compartidos donde personas de diferentes dominios se reúnan para hablar sobre sus avances, las personas que trabajan en un tipo de problema tienen menos probabilidades de exponerse a ideas potencialmente notables en un área diferente.
Claro, muchas ideas sobre economía no se aplicarán a las redes biológicas, pero algunas lo harán, y si los economistas y los biólogos nunca se comunican entre sí, nunca lo sabremos. Si no hay un terreno común, habrá una gran cantidad de reinvención y retrasos, incluso años para que los métodos en un dominio pasen a otro.
Es por eso que creo que es muy importante estudiar y reunirse para debatir sobre las redes en general. Este tipo de fervor interdisciplinario es otra cosa que los físicos y los informáticos ayudaron a poner en marcha hace unos 20 años; eran principalmente físicos y científicos informáticos que transmitían "también podemos hacer sociología, política y ecología".
Esa actitud ciertamente molestó a algunas personas, especialmente a los sociólogos que ya habían estado haciendo redes durante 80 años, pero también generó un enorme y amplio interés en las redes de prácticamente todas las ciencias. Ahora, las diferentes áreas disciplinarias de la ciencia de las redes crecen tan rápido que, de alguna manera, el centro -encrucijada donde las ideas pueden saltar entre los campos- se está reduciendo de manera efectiva.
¿Cómo puede la ciencia de redes fomentar una mayor colaboración entre dominios?
Clauset: Tener un evento real que sirva como una encrucijada entre dominios donde las personas pueden presentarse e interactuar es esencial. En muchos sentidos, la
Conferencia Internacional sobre Ciencia de Redes está tratando de hacer eso, pero se esfuerza por sacar a los investigadores de sus dominios y colocarlos en el medio, ya que las diferentes disciplinas tienen diferentes preguntas generales. Creo que siempre que algunos expertos en dominios de diferentes campos lleguen a la encrucijada para hablar e interactuar, las buenas ideas eventualmente se extenderán.
Continuar con este esfuerzo interdisciplinario será una parte clave para continuar el avance de la ciencia de redes. Pero no todos los esfuerzos deben ser interdisciplinarios. De hecho, las disciplinas son esenciales para ayudar a enfocar nuestra atención colectiva.
No estoy seguro de cuál es el equilibrio correcto entre el trabajo disciplinario y el interdisciplinario, pero para mí las ideas interdisciplinarias son las más interesantes. Si el trabajo sobre estos no está financiado y respaldado a niveles decentes, seguramente no abordaremos muchas de las ideas más importantes de la sociedad porque son las que abarcan diferentes disciplinas.
Por ejemplo, la ciberseguridad no es solo un problema técnico, ya que los humanos tienen un terrible historial de escritura de software libre de errores. La seguridad real requiere componentes legales, componentes sociales, componentes éticos, componentes económicos y probablemente más para desarrollar una solución duradera.
De hecho, si elige cualquier problema que afecte a una porción decente de la población, entonces seguramente se trate de un problema interdisciplinario que requerirá un enfoque interdisciplinario para comprender y resolver.
Conclusión
Como puede ver, tuvimos una gran discusión sobre cómo están cambiando algunas de las ideas preconcebidas sobre las redes. La próxima semana, en la segunda parte de esta serie, resumiré nuestra inmersión más profunda en algunos de los avances y temas emergentes en la ciencia de redes.



 . Muchos estudios de coexpresión utilizan el valor absoluto de la correlación como una medida de similitud de coexpresión sin signo,
. Muchos estudios de coexpresión utilizan el valor absoluto de la correlación como una medida de similitud de coexpresión sin signo,
 y
y  consisten en la expresión de los genes i y j a través de múltiples muestras. Sin embargo, el uso del valor absoluto de la correlación puede ofuscar información biológicamente relevante, ya que no se hace distinción entre la represión de genes y la activación. En contraste, en las redes firmadas, la similitud entre los genes refleja el signo de la correlación de sus perfiles de expresión. Para definir una medida de coexpresión firmada entre los perfiles de expresión génica
consisten en la expresión de los genes i y j a través de múltiples muestras. Sin embargo, el uso del valor absoluto de la correlación puede ofuscar información biológicamente relevante, ya que no se hace distinción entre la represión de genes y la activación. En contraste, en las redes firmadas, la similitud entre los genes refleja el signo de la correlación de sus perfiles de expresión. Para definir una medida de coexpresión firmada entre los perfiles de expresión génica 
 , la similitud firmada
, la similitud firmada  toma un valor entre 0 y 1. Tenga en cuenta que la similitud no firmada entre dos genes expresados de manera opuesta (
toma un valor entre 0 y 1. Tenga en cuenta que la similitud no firmada entre dos genes expresados de manera opuesta ( es igual a 1 mientras que es igual a 0 para la similitud signada. De manera similar, mientras que la medida de coexpresión sin firmar de dos genes con correlación cero permanece en cero, la similitud con signo es igual a 0.5.
es igual a 1 mientras que es igual a 0 para la similitud signada. De manera similar, mientras que la medida de coexpresión sin firmar de dos genes con correlación cero permanece en cero, la similitud con signo es igual a 0.5.![A=[a_{{ij}}]](https://wikimedia.org/api/rest_v1/media/math/render/svg/a1abea4bedfb4eabb36df6d0c0b97aebc10829fb) , se utiliza para cuantificar la fuerza con que los genes están conectados entre sí. A se define mediante el umbral de la matriz de similitud de coexpresión
, se utiliza para cuantificar la fuerza con que los genes están conectados entre sí. A se define mediante el umbral de la matriz de similitud de coexpresión ![S=[s_{{ij}}]](https://wikimedia.org/api/rest_v1/media/math/render/svg/d89ceb0ed7c6f5756f60b1a31f0c6ca8b9891039) . El umbral 'duro' (dicotomización) la medida de similitud S da como resultado una red de coexpresión de genes no ponderada. Específicamente, una adyacencia de red no ponderada se define como 1 si
. El umbral 'duro' (dicotomización) la medida de similitud S da como resultado una red de coexpresión de genes no ponderada. Específicamente, una adyacencia de red no ponderada se define como 1 si  y 0 en caso contrario. Debido a que el umbral difícil codifica las conexiones genéticas de forma binaria, puede ser sensible a la elección del umbral y resultar en la pérdida de información de coexpresión. [3] La naturaleza continua de la información de coexpresión se puede preservar empleando un umbral suave, que da como resultado una red ponderada. Específicamente, WGCNA utiliza la siguiente función de potencia para evaluar la fuerza de su conexión:
y 0 en caso contrario. Debido a que el umbral difícil codifica las conexiones genéticas de forma binaria, puede ser sensible a la elección del umbral y resultar en la pérdida de información de coexpresión. [3] La naturaleza continua de la información de coexpresión se puede preservar empleando un umbral suave, que da como resultado una red ponderada. Específicamente, WGCNA utiliza la siguiente función de potencia para evaluar la fuerza de su conexión: ,
, , la adyacencia de red ponderada está relacionada linealmente con la similitud de coexpresión en una escala logarítmica. Tenga en cuenta que un β de alta potencia transforma altas similitudes en adyacencias altas, mientras que empuja similitudes bajas hacia 0. Dado que este procedimiento de umbral suave aplicado a una matriz de correlación de pares conduce a una matriz de adyacencia ponderada, el análisis que sigue es Se denomina análisis de red de coexpresión de genes ponderados.
, la adyacencia de red ponderada está relacionada linealmente con la similitud de coexpresión en una escala logarítmica. Tenga en cuenta que un β de alta potencia transforma altas similitudes en adyacencias altas, mientras que empuja similitudes bajas hacia 0. Dado que este procedimiento de umbral suave aplicado a una matriz de correlación de pares conduce a una matriz de adyacencia ponderada, el análisis que sigue es Se denomina análisis de red de coexpresión de genes ponderados. , es definido en función de la correlación de cada gen con el respectivo módulo eigengene. El segundo, denominado kIN, se define como una suma de adyacencias con respecto a los genes del módulo. En la práctica, estas dos medidas son equivalentes. [4] Para probar si un módulo se conserva en otro conjunto de datos, se pueden usar varias estadísticas de red, por ejemplo.
, es definido en función de la correlación de cada gen con el respectivo módulo eigengene. El segundo, denominado kIN, se define como una suma de adyacencias con respecto a los genes del módulo. En la práctica, estas dos medidas son equivalentes. [4] Para probar si un módulo se conserva en otro conjunto de datos, se pueden usar varias estadísticas de red, por ejemplo.  . [6]
. [6]











